Mikrobielle Gemeinschaften verraten einiges über den Zustand der Böden. Über diese Biodiversität wird auf genetischer Ebene rund um den Globus sehr viel geforscht, doch nicht immer lassen sich die Forschungsergebnisse von Dritten optimal nutzen.
Der Grund: Die Informationen sind in verschiedenen Datenbanken gesammelt und unterschiedlich gut erfasst. UFZ-Forscher*innen haben nun eine neue Metadaten-Datenbank für terrestrische Metagenome mit mehr als 15.000 Datensätzen aufgebaut, die Wissenschaftler*innen weltweit die Arbeit erleichtern wird. Das schreiben sie im Fachblatt Nucleic Acids Research.
Mehr als 202.000 Metagenome, also jeweils die Gesamtheit der genetischen Information in einer Bodenprobe, finden sich in den beiden wichtigsten Datenbanken, in denen Mikrobiologen weltweit diese Forschungsdaten archivieren können: Die Repositorien MG-Rast und das Sequence Read Archive (SRA). Dort haben Wissenschaftler festgehalten, wo sie mikrobielle Gemeinschaften untersucht oder Genomsequenzierungen gemacht haben und zu welchen Ergebnissen sie gekommen sind.
Damit ermöglichen sie es anderen Forschern, diese Daten für eigene Forschungsfragen zu nutzen und sie mit eigenen Ergebnissen zu vergleichen. Und es erspart ihnen auch, selbst zeitintensive Fragestellungen beantworten zu müssen, für die es vielleicht schon Antworten gibt. Allerdings stoßen die Forscher immer wieder auf Hürden: Die Datensätze sind oft unvollständig und nicht einheitlich beschriftet.
“Dies erschwert es interessierten Anwendern, mit den Daten weiterzuarbeiten”, sagt Dr. Ulisses Nunes da Rocha, Mikrobieller Ökologe am UFZ und einer der Hauptautoren der Studie. Dies fängt bei Kleinigkeiten an, zum Beispiel der Angabe der Temperatur. Sie kann in Fahrenheit, Kelvin oder Celsius erfasst werden, zudem variieren die Schreibweisen für die Abkürzungen dafür.
Doch auch scheinbar Grundsätzliches ist unklar, denn was man beispielsweise genau unter einem Biom (wissenschaftlich für Großlebensraum) versteht, wird von den Wissenschaftlern weltweit durchaus unterschiedlich definiert. All dies, so da Rocha, erschwere es, die Daten effizient zu nutzen.
Aus den Datensätzen von MG Rast und SRA haben Ulisses Nunes da Rocha und sein Team deshalb jene Metagenom-Daten gefiltert, die Wissenschaftler*innen in aller Welt in der terrestrischen Umwelt gesammelt haben. Exakt 15.022 Metagenom-Datensätze aus Wäldern, Grasland und Gesteinen aus 84 Staaten kamen so in der neuen Metadaten-Datenbank zusammen. Für die genaue Beschreibung dieser Metadaten, also beispielsweise die geografischen Koordinaten, den pH-Wert oder die Temperaturen, entwickelten sie keine neuen wissenschaftlichen Standards, sondern nutzten eine bereits bestehende Standardisierungsmethodik.
“Die Metadaten-Datenbank hilft Wissenschaftlern, die auf dem Gebiet der terrestrischen Umwelt forschen und für eigene Fragestellungen mit solchen Daten arbeiten wollen”, sagt der UFZ-Forscher. Anstatt beispielsweise aufwendige Laborexperimente etwa zur CO2-Fixierung oder zur Wirkung von Pestiziden auf mikrobielle Gemeinschaften zu betreiben, können sie in der Datenbank recherchieren, ob nicht schon davor Wissenschaftler anderswo in der Welt dazu vergleichbare Versuche gemacht und ihre Daten zur Verfügung gestellt haben.
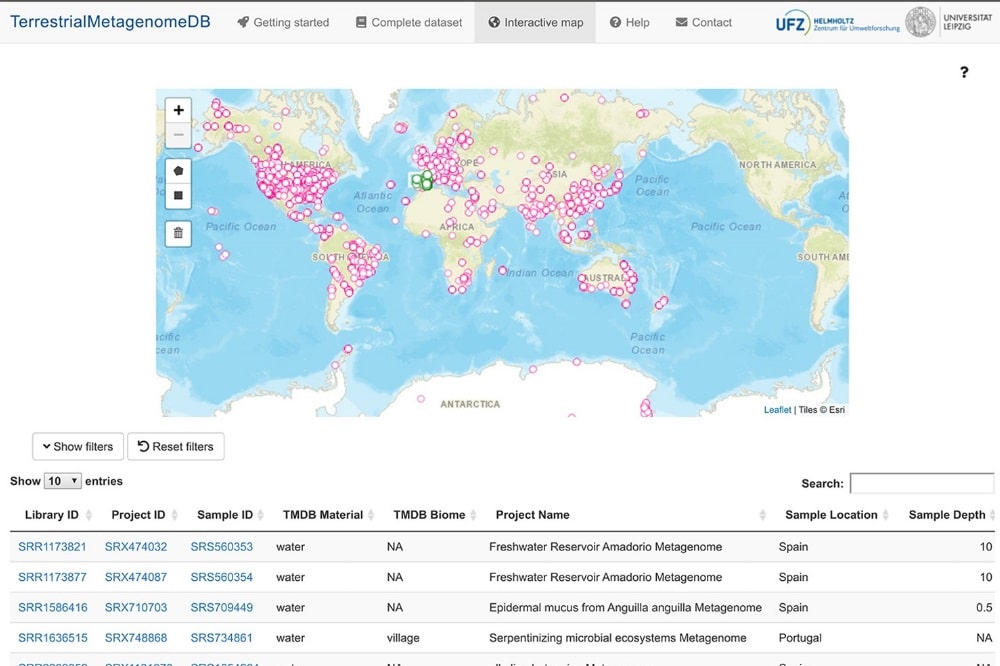
Anfang November ging die frei zugängliche Metadaten-Datenbank unter dem Namen “TerrestrialMetagenomeDB” an den Start. Nutzer*innen können dort über eine Vielzahl von Filtern Daten spezifizieren. Sie können aber auch über eine interaktive Weltkarte Datensätze nach geografischen Gesichtspunkten suchen. Drei Videotutorials unterstützen sie bei der Recherche sowie dem Download der Daten.
Zwei Mal pro Jahr, im Januar und im Juli, wird die Metadaten-Datenbank automatisch aktualisiert. Dabei werden neue oder korrigierte Datensätze von den beiden Repositorien MG-Rast und dem SRA automatisch abgerufen, sofern die Attribute der Daten den Standards der neuen Datenbank entsprechen.
Das Potenzial ist groß, denn dort lagern weitere rund 100.000 Datensätze zu terrestrischen Metagenomen, die bislang nicht standardisiert werden konnten, weil sie nicht sauber erfasst waren. Für Ulisses Nunes da Rocha und seine UFZ-Arbeitsgruppe soll die neue Metadaten-Datenbank der Einstieg sein, um künftig Big Data-Analysen in mikrobiellen Gemeinschaften in terrestrischen Systemen im globalen Maßstab möglich zu machen.
Publikation:
Corrêa, F. B., Saraiva, J. P., Stadler, P. F. & da Rocha, U. N. (2019). TerrestrialMetagenomeDB: a public repository of curated and standardized metadata for terrestrial metagenomes. Nucleic Acids Research, https://doi.org/10.1093/nar/gkz994
Die Datenbank finden Sie unter https://webapp.ufz.de/tmdb/

Keine Kommentare bisher